Wie man mit R schnell auf Analysedaten zugreifen kann
„Ich schau mal eben schnell…“
Der eine oder andere kennt das ja vielleicht: Man hat eine Idee, möchte schnell mal schauen, ob X mit Y zusammenhängt und dann, … ja dann… dann müssen die Daten gesucht, heruntergeladen, importiert und aufbereitet werden – und garantiert geht irgendwo irgendwas schief (meine Lieblinge: seltsame Dezimaltrenner). Kurzum: Man lässt es meistens doch einfach bleiben.
Wie gut, dass es in den letzten Jahren nicht nur immer mehr großartige Datenquellen im Netz gibt, sondern auch fleißige Menschen, die dafür sorgen, dass man mittels R äußert bequem darauf zugreifen kann. Und so kann man dann auch in weniger als 10 Minuten „mal eben schnell schauen“, ob es einen Zusammenhang zwischen den Gesundheitsausgaben eines Landes und der Lebenserwartung in dem Land gibt (ja, okay… Google wäre natürlich noch schneller gewesen, denn zu dieser Frage gibt es hunderte Artikel). Here we go:
Als Datengrundlage dient der Quality of Government Datensatz, der eine Reihe spannender Zeitreihen-Daten auf der Ebene von Nationalstaaten enthält. Vor ein paar Jahren hätte ich mir den Datensatz jetzt halbwegs mühsam im csv-Format heruntergeladen und in R importiert. Glücklicherweise geht das aber auch deutlich einfacher, weil Markus Kainu dankenswerterweise das rqog-package geschrieben hat, welches einen direkten Import der Daten ermöglicht.
# rqog ist nicht auf CRAN gehostet, sondern muss via devtools direkt über github gezogen werden:
# library(devtools)
# install_github("ropengov/rqog")
library(rqog)Über die read_qog-Funktion lässt sich nun der Quality of Government Dataset direkt in R einlesen. In den Argumenten der Funktion lässt sich zudem festlegen, welche „Geschmacksrichtung“ des Datensatzes gewünscht ist (Details dazu hier). Ich verwende hier den Standard-Datensatz des Jahres 2017 in der Zeitreihen-Variante.
qog <- read_qog(which_data = "basic", data_type = "time-series", year = 2017,
data_dir = NULL, file_format = "csv", download_only = FALSE,
cache = TRUE, update_cache = FALSE)
## Local file not found.
## Downloading QoG qog_bas_ts_jan17.csv data
## from http://www.qogdata.pol.gu.se/dataarchive/qog_bas_ts_jan17.csv
## in file: /tmp/Rtmpj5eYZ7/rqog/qog_bas_ts_jan17.csv
## Reading cache file /tmp/Rtmpj5eYZ7/rqog/qog_bas_ts_jan17.csvDas Objekt qog enthält nun den Quality of Government Standard-Zeitreihendatensatz. Für das Jahr 2013 sind alle relevanten Daten vorhanden, deswegen wird mittels dplyr ein separater Datensatz erstellt, der nur Daten aus 2013 enthält. Zudem werden neben den Länderbezeichnungen ausschließlich die Indikatoren zu den Gesundheitsausgaben sowie die Lebenserwartung im Datensatz behalten. Ursprünglich stammen die Daten übrigens aus der World Development Indicators-Datenbank der Weltbank (wdi). Ich nutze die folgenden Indikatoren:
- wdi_exph (Anteil der Gesundheitsausgaben am BIP)
- wdi_exphpr (Anteil der privaten Gesundheitsausgaben am BIP)
- wdi_exphpu (Anteil der öffentlichen Gesundheitsausgaben am BIP)
- wdi_lifexp (Lebenserwartung)
Danach erfolgt die Visualisierung mittels ggplot. Und das Ergebnis? Gar nicht so eindeutig:
library(dplyr)
##
## Attache Paket: 'dplyr'
## The following objects are masked from 'package:stats':
##
## filter, lag
## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
library(ggplot2)
library(ggrepel)
df <- qog %>%
filter(year == 2013) %>%
select(cname, ccodealp, wdi_exph, wdi_exphpr, wdi_exphpu, wdi_lifexp)
# Gesamtausgaben -> Lebenserwartung
ggplot(df, aes(wdi_exph, wdi_lifexp))+
geom_point(size = 2, shape = 20) +
labs(x = "Gesundheitsausgaben (in % am BIP)", y = "Lebenserwartung") +
geom_smooth(method = "auto", alpha=.4, fill="grey", linetype=2, color="black", size = 0.5) +
scale_x_continuous(limits=c(0,18), breaks=seq(0,20,2.5)) +
scale_y_continuous(limits= c(50,90), breaks=seq(0,90,10))
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
## Warning: Removed 34 rows containing non-finite values (stat_smooth).
## Warning: Removed 34 rows containing missing values (geom_point).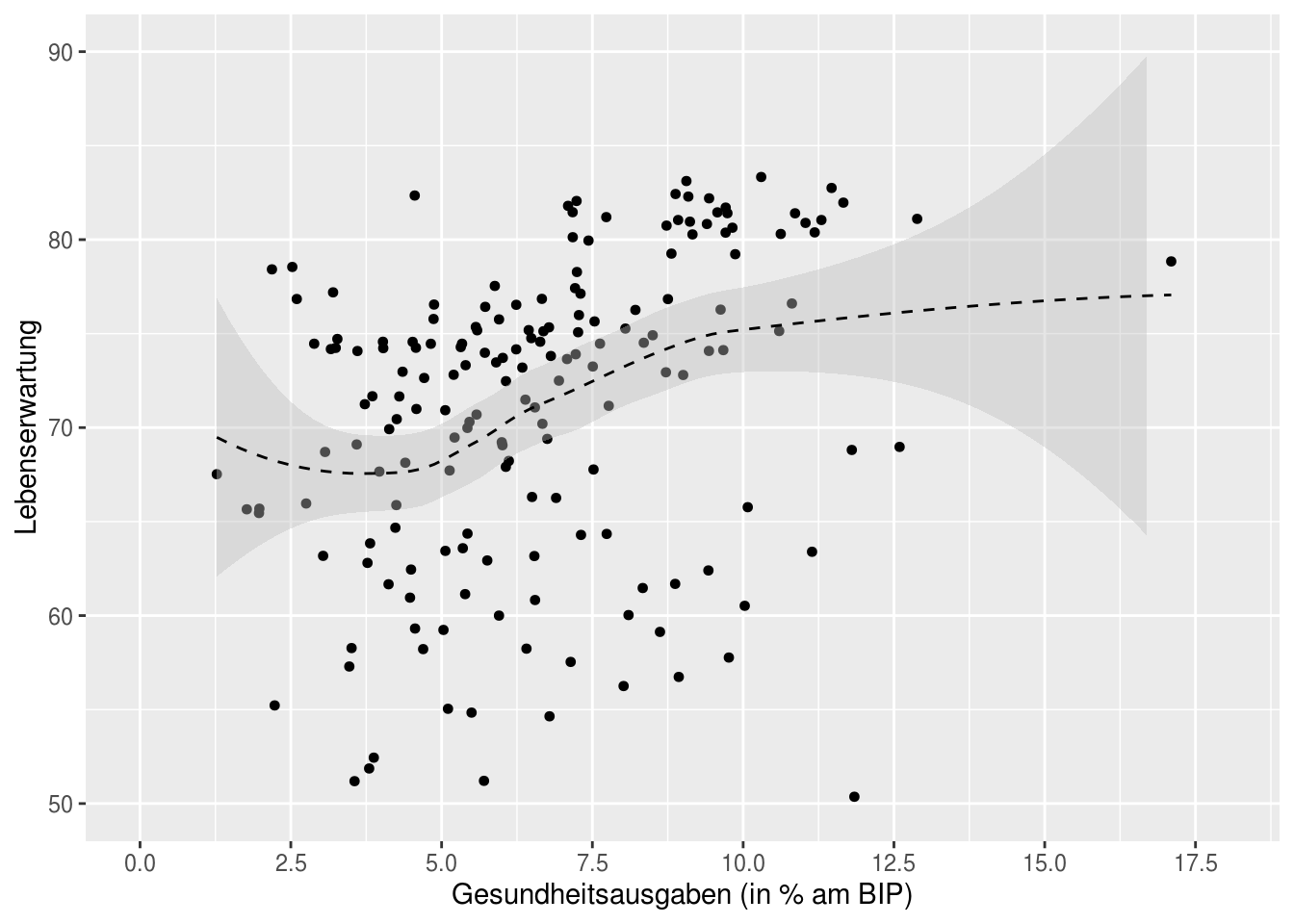
# Private Ausgaben -> Lebenserwartung
ggplot(df, aes(wdi_exphpr, wdi_lifexp))+
geom_point(size = 2, shape = 20) +
labs(x = "Private Gesundheitsausgaben (in % am BIP)", y = "Lebenserwartung") +
geom_smooth(method = "auto", alpha=.4, fill="grey", linetype=2, color="black", size = 0.5) +
scale_x_continuous(limits=c(0,10), breaks=seq(0,20,2.5)) +
scale_y_continuous(limits= c(50,90), breaks=seq(0,90,10))
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
## Warning: Removed 35 rows containing non-finite values (stat_smooth).
## Warning: Removed 35 rows containing missing values (geom_point).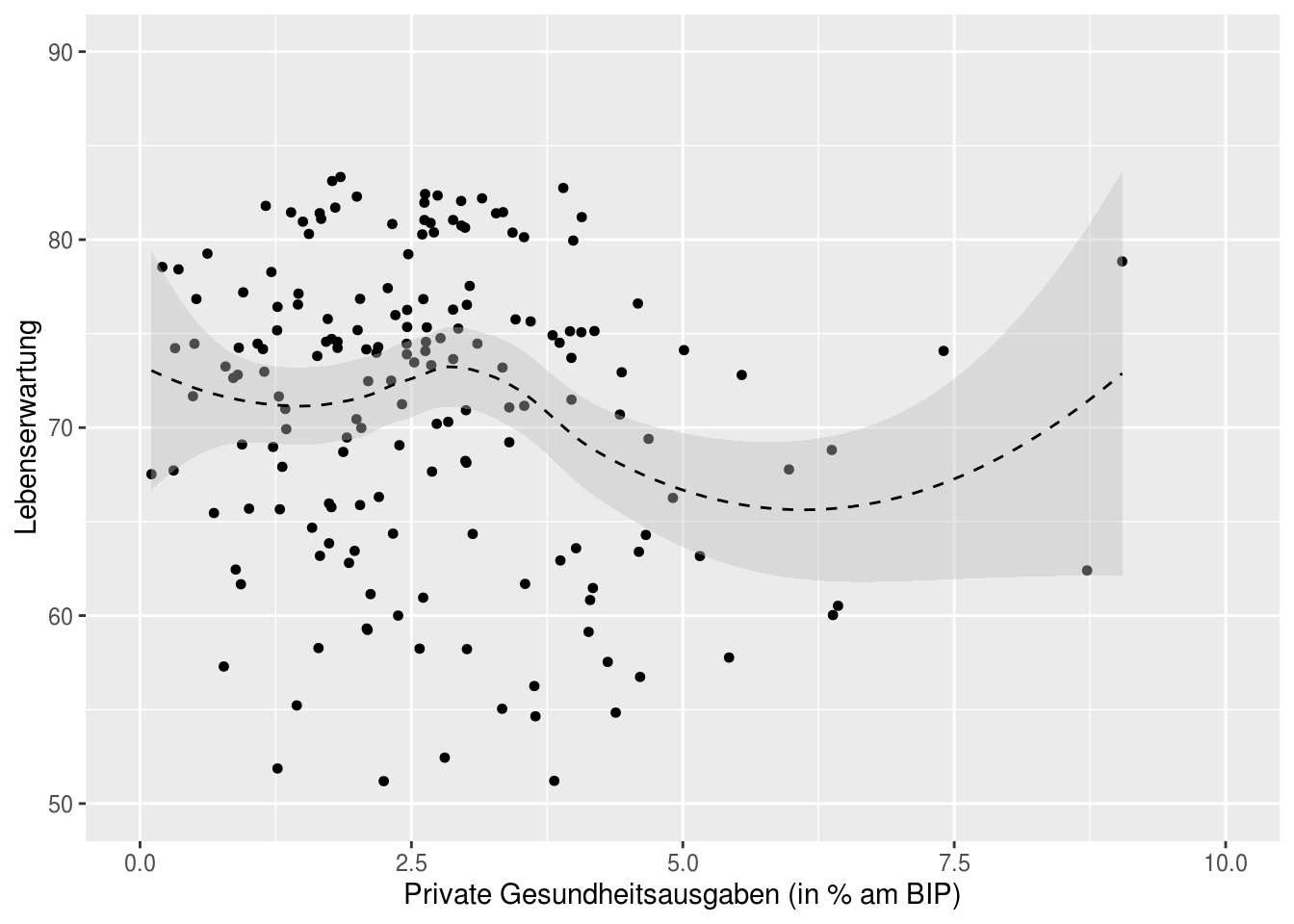
# Öffentliche Ausgaben -> Lebenserwartung
ggplot(df, aes(wdi_exphpu, wdi_lifexp))+
geom_point(size = 2, shape = 20) +
labs(x = "Öffentliche Gesundheitsausgaben (in % am BIP)", y = "Lebenserwartung") +
geom_smooth(method = "auto", alpha=.4, fill="grey", linetype=2, color="black", size = 0.5) +
scale_x_continuous(limits=c(0,11), breaks=seq(0,11,2.5)) +
scale_y_continuous(limits= c(50,90), breaks=seq(0,90,10))
## `geom_smooth()` using method = 'loess' and formula 'y ~ x'
## Warning: Removed 35 rows containing non-finite values (stat_smooth).
## Warning: Removed 35 rows containing missing values (geom_point).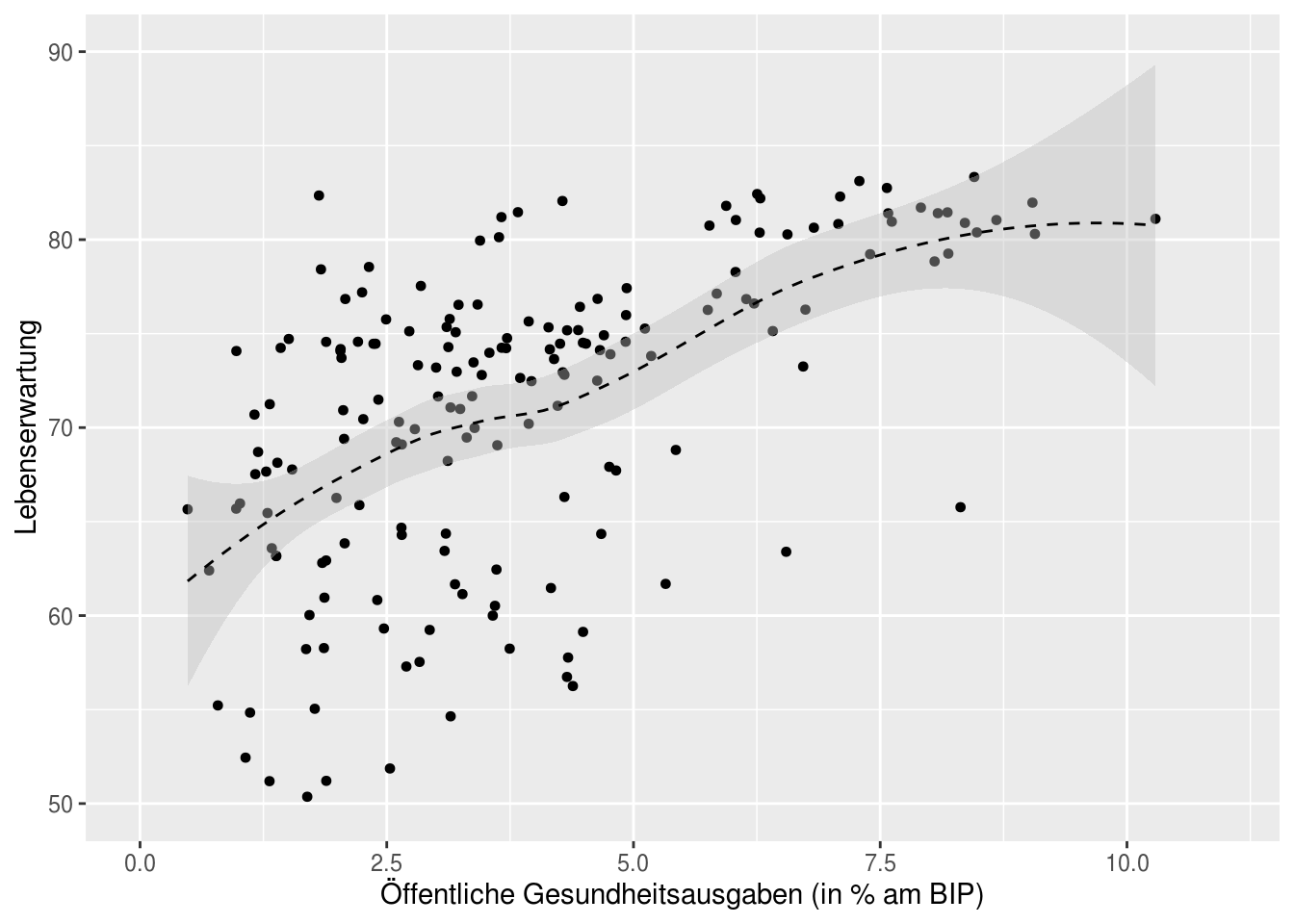
Auf den ersten Blick sind es v.a. die öffentlichen Gesundheitsausgaben, die einen Effekt auf die Lebenserwartung haben. Jetzt müssten natürlich weiterführende Analysen folgen (v.a. müssten Ausreißer identifiziert und kontrolliert werden) – dafür würde ich aber dann sicher nochmal 10 Minuten brauchen…